Newsletter
Genomeditierung mit CRISPR/Cas: Wohin führt der Weg?
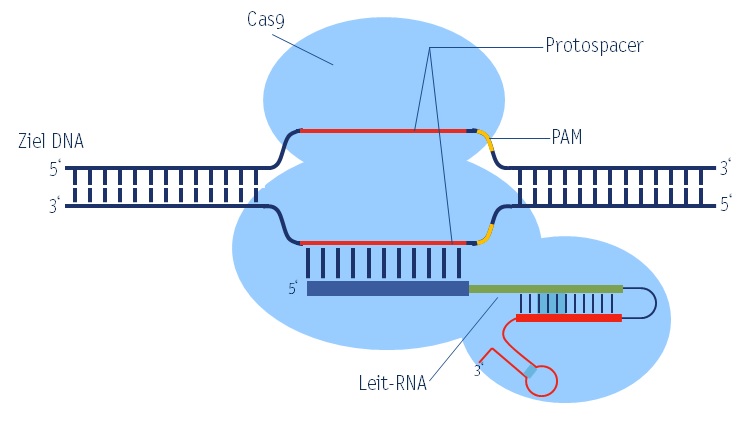
CRISPR/ Cas9, Bild: Karin Garber, Open Science - Lebenswissenschaften im Dialog
Die Genschere CRISPR/Cas stellt eine neue Methode zur Genomeditierung dar und wird als Revolution der Gentechnik gefeiert. Mit ihr kann DNA einfach, schnell und vor allem genau an einer bestimmten Stelle verändert werden, und das in nahezu allen lebenden Zellen und Organismen. Das neue Präzisions-Werkzeug ermöglicht es, DNA gezielt im Genom zu entfernen oder einzufügen. CRISPR/Cas wird mittlerweile standardmäßig in Forschung und Entwicklung eingesetzt und hat maßgeblich dazu beigetragen, dass diese beschleunigt wurden. Vor allem bei der Heilung von Krankheiten und der Züchtung von Pflanzen, die beispielsweise der Klimakrise trotzen sollen, wird große Hoffnung in CRISPR/Cas gesetzt. Bei der Anwendung dieser Methode gibt es allerdings auch ethische Bedenken.
Kleine Geschichte der Genomeditierung: Von der Entdeckung der Restriktionsenzyme bis zur CRISPR/Cas-Methode
Schon lange vor der Entdeckung von CRISPR/Cas war es möglich, gezielte Veränderungen von DNA vorzunehmen. Verschiedene Methoden der Genomeditierung – so nennt man molekularbiologische Techniken zur zielgerichteten Veränderung von DNA – wurden auch zuvor bereits standardmäßig eingesetzt.
Restriktionsenzyme
Der Grundstein dafür, dass DNA gezielt verändert werden kann, wurde 1970 mit der erstmaligen Isolation eines Restriktionsenzyms gelegt. Restriktionsenzyme, auch als Restriktionsendonukleasen bezeichnet, sind Proteine, die bestimmte DNA-Sequenzen erkennen und schneiden können. Restriktionsenzyme sind heute aus dem Labor nicht mehr wegzudenken und werden unter anderem zum Klonieren eingesetzt. Dabei wird ein DNA-Vektor gezielt geschnitten, und an den Schnittstellen werden meistens neue Gene eingefügt. Möchte man jedoch gezielte Änderungen im Genom eines Organismus vornehmen, so gibt es bei der Verwendung von Restriktionsenzymen folgendes Problem: Die Erkennungssequenzen der meisten Restriktionsenzyme sind meist nur wenige Basenpaare lang – bei den Typ II Restriktionsenzymen, die die DNA innerhalb oder in unmittelbarer Nähe der Erkennungssequenz schneiden, sind es in etwa vier bis acht Basenpaare (siehe Tabelle 1). Diese Sequenzen kommen mehrfach im Genom vor, und Restriktionsendonukleasen würden dieses in viele kleine Teile zerstückeln. Restriktionsenzyme sind daher für die Manipulation einer einzigen Region im Genom nicht geeignet.
|
Enzymname |
Erkennungssequenz |
Schnitt |
Enden |
|
|
Eco RI |
5'-GAATTC-3' |
5'-G AATTC-3' |
Sticky |
Überhang mit 4 Basen |
|
3'-CTTAAG-5' |
3'-CTTAA G-5' |
|||
|
Hind III |
5'-AAGCTT-3' |
5'-A AGCTT-3' |
Sticky |
Überhang mit 4 Basen |
|
3'-TTCGAA-5' |
3'-TTCGA A-5' |
|||
|
Bam HI |
5'-GGATCC-3' |
5'-G GATCC-3' |
Sticky |
Überhang mit 4 Basen |
|
3'-CCTAGG-5' |
3'-CCTAG G-5' |
|||
|
Hae III |
5'-GGCC-3' |
5'-GG CC-3' |
Blunt |
Glattes Ende, kein Überhang |
|
3'-CCGG-5' |
3'-CC GG-5' |
|||
|
Sma I |
5'-CCCGGG-3' |
5'-CCC GGG-3' |
Blunt |
Glattes Ende, kein Überhang |
|
3'-GGGCCC-5' |
3'-GGG CCC-5' |
Tabelle 1: Beispiele von Typ II-Restriktionsenzymen und ihren DNA-Erkennungssequenzen. Tabelle von Open Science – Lebenswissenschaften im Dialog (CC BY-SA 3.0 AT)
Artifizielle Nukleasen
In weiterer Folge wurde deshalb an der Entwicklung sogenannter artifizieller Nukleasen gearbeitet, also künstlich hergestellter Proteine, die DNA schneiden können. Ziel war es, die Enzyme genauer zu machen und sie so zu manipulieren, dass sie an längere DNA-Abschnitte binden und so eine Sequenz erkennen, die weniger häufig bzw. im Idealfall nur einmal im Genom vorkommt. Das ist eine wichtige Voraussetzung dafür, um einen gezielten Eingriff an einer bestimmten Stelle im Erbgut vornehmen zu können – wie beispielsweise bei der Gentherapie. Das Prinzip war bei allen eingesetzten Methoden, die daraufhin folgten, stets dasselbe: Endonukleasen schneiden doppelsträngige DNA an einer bestimmten Zielsequenz, es entsteht ein Doppelstrangbruch, und DNA-Reparatur-Prozesse in der Zelle werden in Gang gesetzt. Die DNA wird danach repariert – wodurch auch falsche Sequenzen entstehen können – oder es werden zusätzliche DNA-Elemente eingefügt, die jedoch extra mitgeliefert werden müssen.
Als Resultat der Bemühungen kamen in den 1990er Jahren erstmals so genannte Meganukleasen zum Einsatz. Diese erkennen längere DNA-Sequenzen, sind jedoch immer noch relativ unspezifisch. Bei ihnen ist eine einzige Domäne sowohl für die DNA-Bindung als auch für das Schneiden zuständig. Das Design – also die künstliche Herstellung mit allen damit verbundenen Überlegungen – von Meganukleasen ist sehr anspruchsvoll und zeitintensiv.
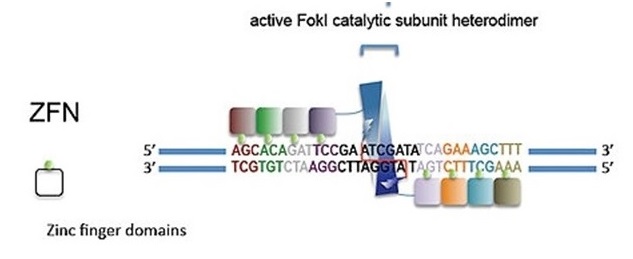
Ebenfalls in den 1990er Jahren gelang es, DNA gezielt mit so genannten Zink-Finger-Nukleasen (ZFN) zu schneiden. Der Trick dabei war folgender: ZFN sind Fusionsproteine und bestehen aus einer Zinkfingerdomäne, die DNA erkennt und bindet, aber nicht schneidet, und einer Nukleasedomäne, die DNA zwar schneiden kann, dies aber nur unspezifisch. Durch die Kombination beider Domänen gelang es nun, die ZNF zielgerichtet an eine bestimmte Stelle der DNA zu bringen und diese dann zu schneiden. Der Nachteil dieser Methode ist jedoch der große Aufwand (mehrere Monate), schwieriges Design, hohe Kosten sowie Off-Target-Effekte (DNA wird auch unspezifisch geschnitten).
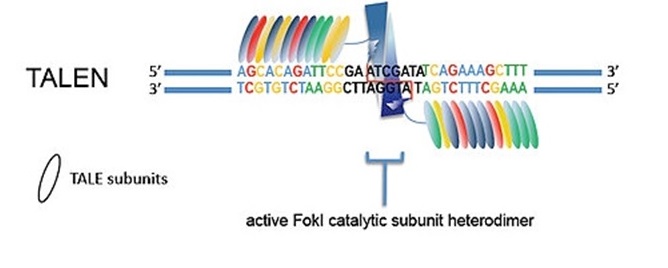
Im Jahr 2010 erfolgte mit dem Einsatz der Transcription Activator-like Effektor Nucleases (TALENS) hier noch eine maßgebliche Verbesserung. Das Prinzip der DNA-Erkennung durch die TALE-Domäne und des Schneidens der DNA durch die Nukleasedomäne blieb dasselbe, der Aufwand konnte jedoch auf wenige Wochen reduziert werden, und insgesamt wurden auch die Kosten gesenkt. TALENS können so designt werden, dass beinahe jede Zielsequenz anvisiert werden kann, das Design ist jedoch schwierig.
Die Genomeditierungsverfahren aus der „Vor-CRISPR-Ära“ hatten somit allesamt erhebliche Nachteile, was auch die Möglichkeiten, Krankheiten durch Gentherapie zu heilen, einschränkte. Das alles änderte sich schlagartig mit der Entdeckung von CRISPR/Cas9: Die „molekulare Genschere“ erlaubt den präzisen, punktgenauen Eingriff in das Erbgut von praktisch jedem Organismus. Diese neue molekularbiologische Methode ist noch dazu leicht anwendbar, schnell und preiswert. Das erklärt, warum sie oft auch als größte Revolution der Molekularbiologie seit der Erfindung der PCR bezeichnet wird.
Entdeckung von CRISPR/Cas9
CRISPR/Cas9 wurde bereits im Jahr 1987 erstmals erwähnt, als es in Bakterien entdeckt wurde [1]. 1993 beschrieb Francisco Mojica den CRISPR-Lokus [2]. Es folgten weitere Publikationen dazu. Erst im Jahr 2012 jedoch legten die französische Forscherin Emmanuelle Charpentier und ihre amerikanische Kollegin Jennifer Doudna mit ihren detaillierten in vitro Studien die Grundlagen für das sogenannte CRISPR/Cas-System [3]. Emmanuelle Charpentier hatte ihre Arbeit zu CRISPR/Cas bereits zuvor im Rahmen des Doktoratskollegs RNA Biologie an den Max Perutz Labs in Wien begonnen, und somit hat die Erfolgsstory der Genschere ihren Ursprung im Prinzip in Wien. CRISPR/Cas9 wurde ursprünglich als Teil des adaptiven Immunsystems von Bakterien zur Abwehr von Viren entdeckt und für die Verwendung in vitro – also außerhalb des lebenden Organismus – beschrieben. Bald jedoch gelang es ForscherInnen auf der ganzen Welt, die DNA verschiedenster Organismen damit zu verändern: CRISPR/Cas9 funktioniert in Einzellern, Pilzen, Pflanzen, Tieren und auch beim Menschen.
CRISPR/Cas9 – Abwehrsystem von Bakterien
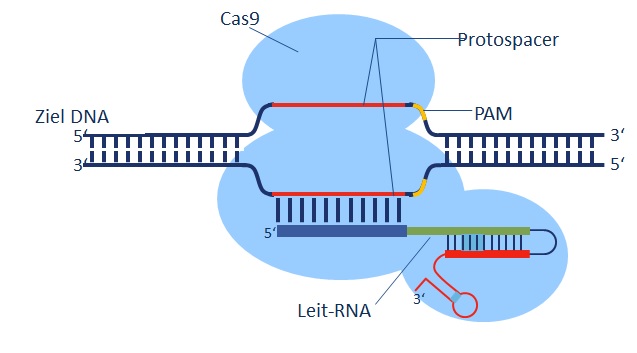
CRISPRsteht für „clustered regularly interspaced short palindromic repeats“ und bezeichnet Regionen im Genom, in denen sich DNA-Sequenzen wiederholen. In der Nähe der CRISPR-Sequenzen befinden sich sogenannte Cas-Gene, die für wichtige Enzyme des bakteriellen Abwehrsystems kodieren. Eines davon ist die Nuklease Cas9 (CRISPR associated protein 9), ein Enzym, das doppelsträngige DNA schneidet. Meist wird bei Anwendungen des CRISPR/Cas-Systems heute Cas9 von Staphylococcus aureus verwendet. Abbildung 3 gibt einen Überblick über die molekularen Abläufe im CRISPR/Cas-System.
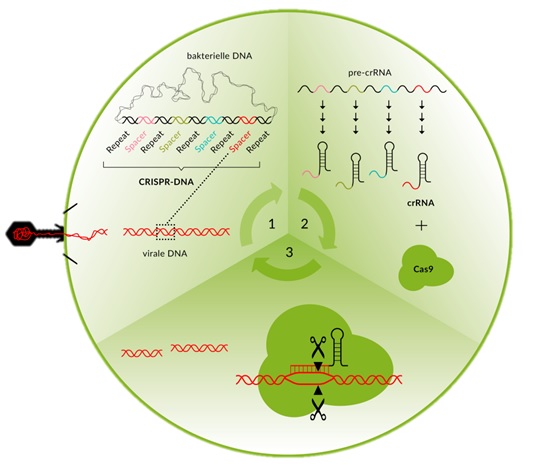
Werden Bakterien von Viren (Bakteriophagen) befallen, so injizieren diese ihre Nukleinsäure ins Bakterium. Die Bakterien reagieren mit der Produktion von Cas9, das Stücke aus der viralen Nukleinsäure herausschneidet und an den CRISPR-Loci ins eigene (Bakterien-) Genom integriert. Bei erneuter Attacke der gleichen Viren werden die CRISPR-Loci, die nun auch die viralen DNA-Sequenzen enthalten, in kurze RNA-Moleküle (crRNA und trRNA) kopiert. Diese binden gemeinsam dann an Cas9. Die RNA-Enzym-Komplexe (crRNA-trRNA-Cas9) erkennen invasive virale Nukleinsäuren über ihre RNA-Bestandteile, die passende homologe Sequenzen in der Virus-DNA finden und das Cas9-Protein quasi dorthin lotsen. Cas9 bindet so an den entsprechenden DNA-Bereich und schneidet die Zielsequenz. Dies geschieht aber nur dann, wenn diese in Kombination mit einem sogenannten PAM-Motiv (Protospacer adjacent motif), einer 2-6 bp langen DNA-Sequenz, die unmittelbar der Cas9-Zielsequenz folgt, vorliegt. Das Virus wird so daran gehindert, sich im Wirtsbakterium zu vermehren. Da die CRISPR-Sequenzen des Bakteriums keine PAM-Motive enthalten, wird die bakterielle DNA nicht von Cas9 geschnitten.
Im Labor wird CRISPR/Cas9 mittlerweile standardmäßig angewandt, um zielgenau DNA zu verändern. Hierbei kommen außer Cas9 auch andere Cas-Proteine, wie Cpf1, Cas12 oder Cas13, zum Einsatz. Diese haben im Vergleich zu Cas9 leicht abweichenden Eigenschaften - Cas13 wird beispielsweise bei RNA-Viren in Pflanzen verwendet. Die Bauteile der crRNA und trRNA werden in der molekularbiologischen Anwendung zu einer sogenannten guideRNA (gRNA) fusioniert. In der gRNA wird die crRNA von den ForscherInnen so designt, dass sie die Ziel-DNA binden kann. Je nach verwendetem Cas-Protein muss beim Design der gRNA darauf geachtet werden, dass in der Nähe der Ziel-DNA die richtige PAM-Sequenz vorhanden ist.
Anwendungen von CRISPR/Cas
Die CRISPR/Cas Technologie hat zuvor undenkbare Möglichkeiten in Forschung und Entwicklung eröffnet, und der Einsatz der Genschere boomt weltweit. Wurde anfangs nur in Zellen und Versuchstieren damit experimentiert, so gibt es mittlerweile auch schon vereinzelte Anwendungen am Menschen. Die Anzahl der veröffentlichten Studien zu CRISPR/Cas sowie Patentanmeldungen, die damit zusammenhängen, sind im Laufe der letzten Jahre rasant gestiegen. [4]
Im Prinzip wird CRISPR/Cas heute für drei große Bereiche eingesetzt: Medizin, Landwirtschaft und Grundlagenforschung. Hier ein paar ausgewählte Beispiele aus den vielen Erfolgen, die mit der Genschere schon verzeichnet werden konnten:
Medizin
- CRISPR-Therapie bei Erkrankungen des Blutes: Es gibt bereits mehrere Ansätze, um die Sichelzellkrankheit mithilfe der CRISPR/Cas-Technologie zu heilen. Bei einer der Methoden werden Blutstammzellen von PatientInnen aus deren Knochenmark gewonnen. Mithilfe von CRISPR/Cas9 und einer korrekten DNA-Vorlage wird der fehlerhafte Baustein in der DNA korrigiert. Die defekten Stammzellen im Körper der Patientin/des Patienten werden durch Chemotherapie zerstört, und die korrigierten Stammzellen werden mittels Transfusion in den Blutkreislauf gebracht. Diese Methode konnte am Menschen bereits erfolgreich angewandt werden. Auch eine Patientin mit Beta-Thalassämie konnte mittlerweile mithilfe von CRISPR/Cas geheilt werden. Sowohl die Behandlung der Sichelzellkrankheit als auch die von Beta-Thalassämie erfolgte allerdings noch im Rahmen von klinischen Studien, für eine standardmäßige Anwendung fehlen aktuell noch statistisch signifikante Aussagen über Wirksamkeit und Risiken.
- CRISPR-Therapie bei Muskeldystrophie: Sowohl bei Mäusen als auch bei Hunden konnte der Defekt im Dystrophin-Gen (Muskelstrukturprotein, das bei Duchenne-Muskeldystrophie betroffen ist) bereits mit Hilfe von CRISPR/Cas9 korrigiert werden. Die Herausforderungen bestehen aktuell noch darin, die Immunantwort des Organismus gegen Cas9 zu unterdrücken und unspezifische und nicht gewollte Veränderungen (Off-Target-Effekte) zu verringern bzw. auszuschließen. Außerdem sind viele verschiedene Muskelarten und Bereiche im Körper betroffen, was die zielgenaue Therapie erschwert.
Landwirtschaft (Nutzpflanzen, Biotechnologie, Sonstiges)
- Krankheitsresistente Kakaopflanzen durch CRISPR/Cas: Die Kakaopflanze (Theabroma cacao)wird oft von Pilzen, Viren oder Schädlingen befallen, und dadurch geht häufig mehr als ein Fünftel der Jahresernte verloren. Die so genannte Schwarzfäule (black pod) ist ein Beispiel einer solchen Pilzkrankheit. Die Kakaopflanze wird bei Befall landwirtschaftlich unbrauchbar, die Folge sind Existenzschwierigkeiten für Landwirte. Die Blätter der Kakaopflanze konnten mithilfe von CRISPR/Cas so verändert werden, dass sie deutlich resistenter gegen Krankheitserreger sind. CRISPR/Cas soll auch dabei helfen, den Kakaopflanzen Insektenresistenz zu verschaffen und ihnen die Anpassung an Klimaveränderungen zu erleichtern. Zurzeit wird die Modifikation von Kakaopflanzen erforscht und ist noch nicht in der Anwendung.
- CRISPR/Cas gegen braunes Obst und Gemüse: Äpfel, Kartoffeln oder Pilze werden nach dem Aufschneiden durch Reaktion mit Luftsauerstoff braun. Dafür sind Polyphenole und das Enzym Polyphenoloxidase (PPO) verantwortlich. Diese sind normalerweise voneinander isoliert, kommen aber durch das Aufschneiden (Zerstören der Zellmembranen) in Kontakt und reagieren mit Luftsauerstoff zu Chinon (gelb) und Melanin (braun). Beim Zuchtchampignon Agaricus bisporus wird CRISPR/Cas dafür eingesetzt, die PPO-Gene dauerhaft zu zerstören. Dadurch wird die Enzym- und damit die Bräunungsaktivität um 30 % reduziert. Nach der europäischen Gentechnik-Richtlinie wäre diese Champignonart jedoch zulassungspflichtig (siehe auch gesetzliche Regelungen, nächster Abschnitt).
- Virenresistente Joghurtkulturen mit CRISPR/Cas: Das Bakterium Streptococcus thermophilus fermentiert Laktase (Milchzucker) zu Milchsäure. Dies wird bei der Joghurtproduktion eingesetzt und führt zur gallertartigen Textur und zum charakteristischen Geschmack von Joghurt. Joghurtbakterien können von sogenannten Bakteriophagen (Viren, die Bakterien infizieren) befallen werden, was Probleme für die Joghurtindustrie bringt. Mithilfe des CRISPR/Cas-Systems konnten phagenresistente Joghurt-Bakterien der Art Streptococcus thermophilus hergestellt werden, die eindringende Phagen effizient erkennen und ihre DNA zerstören.
- CRISPR/Cas für Gene-Drive gegen Malaria: Die Gene-Drive-Technik ist ein „Turbo für die Vererbung“ und beschleunigt die Ausbreitung eines (manipulierten) Gens, indem sich dieses selbstständig auf das Schwesternchromosom kopiert. Eine bestimmte Genvariante wird so nahezu an 100 % aller Nachkommen weitergegeben, und auch bei Selektionsnachteil wird deren Verbreitung in einer gesamten Population oder Art ermöglicht. Das CRISPR/Cas-System wird für den Gene Drive veränderter Gene eingesetzt: Mit CRISPR/Cas werden Mutationen gemeinsam mit einem speziellen Kopiermechanismus in einen Organismus eingebracht und werden dadurch von Generation zu Generation an alle Nachkommen weitervererbt. Hier gibt es beispielweise bereits Ansätze, die weiblichen Moskitos der Gattung Anopheles – Überträger der Malaria – mit der CRISPR/Cas-Methode so zu verändern, dass sie unfruchtbar werden. So gelang es im Labor schon, Populationen von Anopheles gambiae nach nur sieben bis elf Generationen auszurotten [5]. Solche Eingriffe könnten in der Natur jedoch die ursprüngliche Art in kürzester Zeit verändern und unvorhersehbare Auswirkungen auf das Ökosystem haben. Daher ist es wichtig, Gene-Drive nur in gut gesicherten Laboren und Probeumgebungen zu untersuchen, um die Auswirkungen diese Methode besser verstehen zu können.
Grundlagenforschung
- CRISPR/Cas-Fluoreszenzmarkierungen: Um die Funktion bestimmter Zellen besser zu verstehen, werden in der Grundlagenforschung häufig Bestandteile lebender Zellen sichtbar gemacht. Dafür werden unter anderem Fluoreszenzfarbstoffe eingesetzt, die unter speziellen Mikroskopen in einer definierten Farbe leuchten. Mithilfe von CRISPR/Cas können relativ rasch fluoreszierende (Fusions-)Proteine generiert werden, indem ein Fluoreszenzgen in einen definierten DNA-Abschnitt eingebaut wird. Eine andere Anwendung ist das Markieren der Position eines Gens auf dem Chromosom (Genlokus) in der lebenden Zelle. Dafür wird eine inaktivierte Form von Cas9 verwendet („death“ Cas9, dCas9), die mit GFP (green fluorescent protein) verbunden ist. Das fluoreszierende dCas9-GFP-Fusionsproteins wird über die gRNA an einen bestimmten Genlokus gebracht und bindet diesen. Der genaue Ort, wo sich dieser befindet, wird durch die Fluoreszenz ersichtlich.
Gesetzliche Regelungen
Veränderungen durch CRISPR/Cas, bei denen keine Fremd-DNA eingefügt wird (kein Transgen erzeugt wird), könnten auch auf natürliche Weise entstehen und sind nicht nachweisbar. Ob durch CRISPR modifizierte Organismen rechtlich als genetisch veränderte Organismen einzustufen sind oder nicht, wurde daher intensiv diskutiert.
Im Juli 2018 erfolgte dazu ein Urteil des Europäischen Gerichtshofs (EuGH): Neuere Methoden der Gentechnik, die keine transgenen Organismen erzeugen, fallen in Europa trotzdem unter die bestehenden Gentechnik-Richtlinien und gelten rechtlich als gentechnisch veränderte Organismen (GMO, genetically modified organisms). Sie unterliegen strengsten Auflagen, müssen langwierige und teure Zulassungsverfahren durchlaufen und gekennzeichnet werden. Konventionelle Mutageneseverfahren (chemisch, mit Strahlung) hingegen sind von dieser Regelung ausgenommen und die so entwickelten Pflanzen sind nicht als GVO einzustufen, weshalb das strenge Urteil des EuGH für viele überraschend kam. Sein restriktives Urteil begründete der EuGH mit dem Schutz der menschlichen Gesundheit und dem Vorsorgeprinzip: Man müsse den Risiken, die sich aus der Freisetzung gentechnisch veränderter Organismen (GVO) in die Umwelt ergeben, besondere Aufmerksamkeit schenken und vorbeugende Maßnahmen ergreifen.
In Österreich ist der Anbau von gentechnisch veränderten Pflanzen generell verboten. Anwendungen am Menschen (z.B. somatische Gentherapie) sind bewilligungspflichtig.
Ähnlich restriktiv wie die EU wird der Einsatz von CRISPR/Cas beispielsweise auch in Neuseeland gehandhabt. Weniger streng sind hingegen die Schweiz, Norwegen sowie die USA. In den USA sind CRISPR-Lebensmittel zum Verkauf zugelassen, und geneditierte Pflanzen ohne Fremd-DNA sind vom Gentechnikgesetz ausgenommen. Diese unterschiedlichen Regelungen in den verschiedenen Ländern führen jedoch zu folgendem Dilemma: Bestimmte Eingriffe mit CRISPR/Cas sind in der EU zwar verboten, können bei Importen jedoch nicht nachgewiesen werden.
Es erfolgte daher bereits auch die Kritik vieler europäischer ForscherInnen, die sie im Juli 2019 in einem Positionspapier zusammenfassten [6]. Viele WissenschaftlerInnen äußerten sich besorgt, da die strenge EU-Regelung Feldstudien und Nutzpflanzenzüchtungen mithilfe von Genom-Editierung praktisch unmöglich macht.
Beim Eingriff ins Erbgut muss generell zwischen einer Veränderung in somatischen Zellen und in Keimzellen unterschieden werden. Bei der so genannten somatischen Gentherapie wird ins Genom von Körperzellen eingegriffen, um genetisch bedingte Erkrankungen zu lindern oder zu heilen. Die in der DNA eingefügten Veränderungen werden nicht an die Nachkommen vererbt und betreffen nur das behandelte Individuum selbst. Bei der sogenannten Keimbahn-Therapie werden Veränderungen an Keimbahnzellen, Keimzellen oder frühen Embryonen durchgeführt und auch an die Nachkommen weitergegeben.
Bei den heute bekannten Formen der Gentherapie ist eine Manipulation der Keimbahn meist nicht notwendig. Daher gab es auch große Empörung in der Wissenschafts-Community, als ein chinesischer Forscher 2018 behauptete, Gene von zwei Babys mithilfe von CRISPR/Cas in der Keimbahn so manipuliert zu haben, dass diese vor Aids geschützt seien. Die Österreichische Akademie der Wissenschaften veröffentlichte in Reaktion darauf eine Stellungnahme dazu, in der sie zur Anwendung von CRISPR/Cas am Menschen folgende Meinung vertrat: CRISPR/Cas-Editierung der Keimbahn ist als Gentherapie aktuell nicht vertretbar, und Human Enhancement (Verbesserung des menschlichen Körpers) als Ziel von Genom-Editierung ist niemals vertretbar [7]. Der Deutsche Ethikrat plädierte in einer Stellungnahme 2019 für einen globalen Konsens über ethische Mindestanforderungen [8].
Neueste Entwicklungen und Zukunftsvisionen
Die Entdeckung von CRISPR/Cas9 löste einen wahren Hype aus, und die neue Technologie ist mittlerweile weltweit aus Forschung und Entwicklung nicht mehr wegzudenken. Die Genschere ist heute nicht mehr auf Cas9 limitiert, es werden auch andere Cas-Proteine mit leicht unterschiedlichen Eigenschaften eingesetzt. Forschung und Entwicklung haben durch CRISPR/Cas enorm an Geschwindigkeit zugelegt. Versuche und Studien, die vor wenigen Jahren noch viel Zeit und Geld in Anspruch genommen hätten, können heute dank der Genschere schneller und günstiger durchgeführt werden.
CRISPR/Cas wird seit seiner Entdeckung nicht nur in den verschiedensten Bereichen eingesetzt, auch an der Methode selbst wird seitdem noch getüftelt.
- Heute ermöglicht es das sogenannte Prime Editing, einen noch präziserenund flexibleren Eingriff ins Erbgut vorzunehmen. Auch hier kommt eine RNA zum Auffinden der Ziel-DNA zum Einsatz, es wird dabei allerdings auch gleich die Vorlage für die gewünschte Veränderung mitgeschickt. Der DNA-Doppelstrang wird beim Prime Editing nicht durchtrennt. Stattdessen dient die RNA-Vorlage dazu, den bisherigen Abschnitt im Genom mittels Reverser Transkriptase – einem Enzym, das die Umschreibung von RNA in DNA katalysiert – gegen die gewünschte Sequenz auszutauschen [7].
- CRISPR-Switch hat der CRISPR/Cas-Technologie einen weiteren Feinschliff verpasst: Die am Wiener Institut für Molekulare Pathologie etablierte Methode ist ein „Upgrade“ der Genschere und erlaubt ihr gezieltes An- und Ausschalten [8]. Dies wird dadurch erreicht, dass die Aktivität der guideRNAs unter die Kontrolle von Cre-Rekombinasen gebracht wird. Dadurch wird unter anderem ein Ausschalten von Genen in definierter zeitlicher Reihenfolge möglich, was gerade für die Untersuchung komplexer Krankheiten wichtig ist.
Es ist zu erwarten, dass CRISPR/Cas in Zukunft auch weiterhin starken Einfluss auf Anwendungen in den Bereichen Medizin, Tier- und Pflanzenzüchtung sowie Biotechnologie haben wird. Auch die Grundlagenforschung wird dadurch weiterhin mit zuvor nicht denkbarer Geschwindigkeit vorangetrieben werden. Große Hoffnung wird aber nicht nur im medizinischen Bereich in die Genschere gesetzt. CRISPR/Cas könnte in Zeiten der fortschreitenden Klimakrise auch bei der Züchtung von Pflanzen und Tieren zum Einsatz kommen und ihnen ein Fortbestehen auch bei höheren Temperaturen erlauben. Wo uns der Weg mit CRISPR dann wirklich hinführt, kann aktuell – auch aufgrund der rasanten Entwicklung auf diesem Feld – niemand wirklich voraussagen.
as, 16.12.2019
Quellenangaben
[1] Ishino Y., Shinagawa H., Makino k. et al.: Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product (1987). J Bacteriol, 169 (12), 5429-33, Dec 1987. DOI: 10.1128/jb.169.12.5429-5433.1987
[2] Mojica FJM, Juez G. and Rodriguez‐Valera F.: Transcription at different salinities of Haloferax mediterranei sequences adjacent to partially modified PstI sites (1993). Molecular Microbiology, Volume9, Issue3, August 1993, Pages 613-621
[3] Jinek M., Chylinski K., Fonfara I.: A Programmable Dual-RNA–Guided DNA Endonuclease in Adaptive Bacterial Immunity (2012). Science 17 Aug 2012, Vol. 337, Issue 6096, pp. 816-821, DOI: 10.1126/science.1225829
[4] Statistik zu Patentanmeldungen im Zusammenhang mit CRISPR/Cas
[5] Kyrou K., Hammond AM, Galizi R. et al.: A CRISPR–Cas9 gene drive targeting doublesex causes complete population suppression in caged Anopheles gambiae mosquitoes (2018). Nature Biotechnology volume 36, pages1062–1066(2018).
[6] Max Planck Gesellschaft: Stellungnahme zu den wissenschaftlichen und translationalen Auswirkungen der Genom-Editierung und daraus resultierenden ethischen, rechtlichen und gesellschaftlichen Fragen (2019). Positionspapier.
[7] Österreichische Akademie der Wissenschaften: Stellungnahme der Österreichischen Akademie der Wissenschaften zur Genom-Editierung angesichts der jüngsten Berichte über geneditierte Kinder (2018).
[8] Deutscher Ethikrat: Eingriffe in die menschliche Keimbahn. Stellungnahme (2019).
[9] Anzalone AV, Randolph PB, Davis JR et al.:Search-and-replace genome editing without double-strand breaks or donor DANN (2019). Nature volume 576, pages149–157(2019)
[10] Chylinski K., Hubmann M., Hanna RE et al.: CRISPR-Switch regulates sgRNA activity by Cre recombination for sequential editing of two loci (2019). Nature Communications volume 10, Article number: 5454 (2019), DOI 10.1038/s41467-019-13403-y
Weiterführendes zum Thema CRISPR/Cas von Open Science:
- Webinar „Genom-Editierung & die Genschere CRISPR/Cas“ mit Renée Schroeder (Video-Mitschnitt sowie Folien zum Download)
- Video-Interview "Die Genschere CRISPR/Cas - Auf dem Weg zur großen Gen-Revolution?"mit Stefan Ameres
- Aufgabenblatt für den Biologie-Unterricht (Oberstufe) zur Genschere CRISPR/Cas
- Erklärvideo zur Genschere CRISPR/Cas
- Foliensatz zur Genschere CRISPR/Cas
- CRISPR/Cas-Quiz
- CRISPR/Cas-Kreuzworträtsel
- Interviews mit Wissenschaftlern (Christoph Bock, Ulrich Elling und Andreas Bachmair)